Un correcto asesoramiento genético resulta fundamental para explicar, de manera clara y concisa, las implicaciones de un resultado genético. Esto incluye la explicación de los resultados y la implicación que tienen tanto para el paciente como para sus familiares.
Pero, ¿Cómo se clasifican las variantes genéticas que informamos a los pacientes? Es una de las preguntas más recurrentes en las consultas de genética clínica. En esta entrada, entenderemos un poco más acerca de como se realiza este proceso.
Actualmente, las nuevas técnicas genómicas para la realización de estudios genéticos, permite estudiar a la vez desde unos pocos genes (panel), todos los genes (exoma) o incluso todo el genoma completo (genoma). Esto ha provocado que se detecten muchas variantes, y con ello la importancia de su clasificación y su asociación con la patología por la que consulta el paciente.
Por ello, el colegio americano de genetistas médicos y genómicos (ACMG), han elaboras unas guías que han permitido unificar y estandarizar, la clasificación de variantes genéticas una vez se detectan, y su asociación con una enfermedad genética. Estas guías se han adoptado prácticamente a nivel internacional, por cualquier centro que se dedica al diagnóstico genético como guía estándar de clasificación de variantes.
Para ello, establecieron 5 niveles de evidencia para cuando se detecta una variante, en función de su posible asociación con una enfermedad genética. Así, una variante se puede clasificar como benigna, probablemente benigna, variante de significado incierto (VUS), probablemente patogénica y patogénica. Aquellas variantes clasificadas como benignas y probablemente benignas, se considera que no explicarían el fenotipo del paciente, las variantes patogénicas y probablemente patogénicas se considera que están asociadas con una patología genética y las variantes llamadas de significado incierto, se considera que no existe suficiente información para confirmar o descartar el efecto de esa variante.
Para clasificar una determinada variante en el sistema de 5 niveles cualitativos, la ACMG sugiere la revisión de una serie de criterios que permiten la categorización de las variantes en función de su efecto, localización dentro de los dominios de la proteína codificada, su frecuencia en la población general, el patrón de herencia, la co-segregación de la variante con la enfermedad, predicción de patogenicidad por predictores bioinformáticos y el análisis de datos funcionales (in vitro e in vivo). En la figura 1. Se describen los criterios que se tienen en cuenta para la clasificación de una variante según la ACMG.
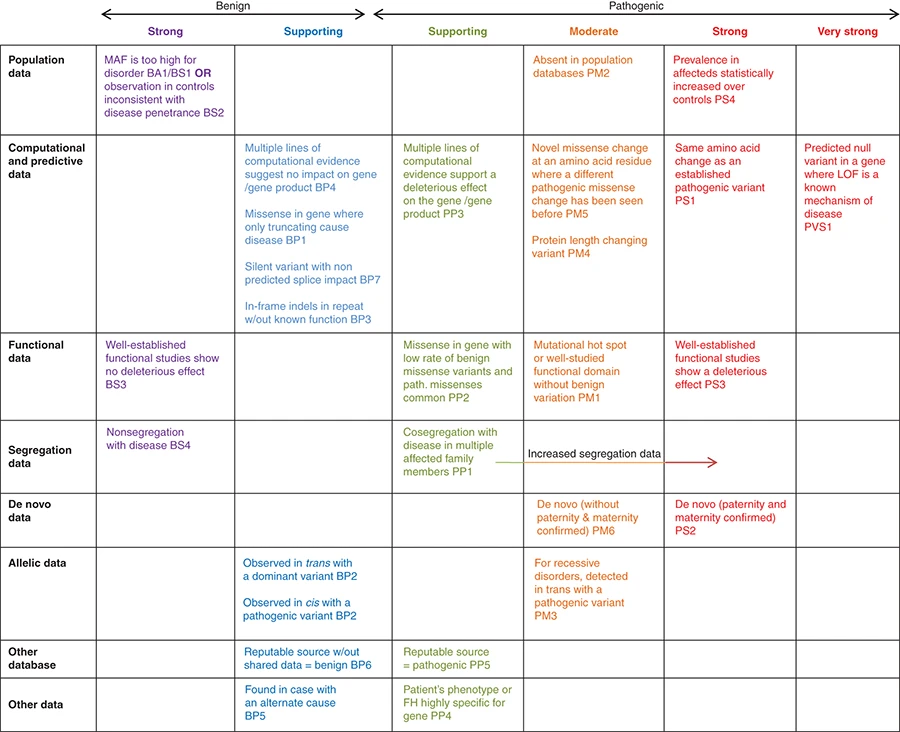
Figura 1. Criterios de clasificación de variantes de la ACMG1
Las variantes que finalmente se clasifican como de significado incierto, son aquellas que generan mayor incertidumbre, ya que no podemos asociarlas ni descartarlas con una determinada enfermedad, y en esos casos será necesario obtener más información (segregación de la variante, estudio funcional, etc.)
Para la ACMG, independientemente del estudio genético que se realice, principalmente exoma y genoma, para diagnóstico, siempre se deben analizar una lista de genes que se llaman “accionables” y en los cuales, la identificación de una variante genética, tendrá una acción en el paciente, en forma de seguimiento clínico, tratamiento y/o cualquier intervención médica que pueda mejorar el pronóstico del paciente. Actualmente, 73 genes forman el listado de genes accionables de la ACMG.
Del mismo modo, la ACMG ha elaborado guías para la clasificación de variantes de numero de copia o CNVs2 y variantes en cáncer, llamadas también guías AMP3
Por ello, es necesario un correcto asesoramiento genético y un correcto análisis de las variantes detectadas, que permita una correcta clasificación e implicación en las enfermedades genéticas.
Lecturas recomendadas:
- Richards, S., Aziz, N., Bale, S. et al. Standards and guidelines for the interpretation of sequence variants: a joint consensus recommendation of the American College of Medical Genetics and Genomics and the Association for Molecular Pathology. Genet Med 17, 405–423 (2015). https://doi.org/10.1038/gim.2015.30
- Riggs ER, Andersen EF, Cherry AM, et al. Technical standards for the interpretation and reporting of constitutional copy-number variants: a joint consensus recommendation of the American College of Medical Genetics and Genomics (ACMG) and the Clinical Genome Resource (ClinGen) [published correction appears in Genet Med. 2021 Nov;23(11):2230]. Genet Med. 2020;22(2):245-257. doi:10.1038/s41436-019-0686-8
- Li MM, Datto M, Duncavage EJ, Kulkarni S, Lindeman NI, Roy S, Tsimberidou AM, Vnencak-Jones CL, Wolff DJ, Younes A, Nikiforova MN. Standards and Guidelines for the Interpretation and Reporting of Sequence Variants in Cancer: A Joint Consensus Recommendation of the Association for Molecular Pathology, American Society of Clinical Oncology, and College of American Pathologists. J Mol Diagn. 2017 Jan;19(1):4-23. doi: 10.1016/j.jmoldx.2016.10.002. PMID: 27993330; PMCID: PMC5707196.

Leave a Reply